Главная
/
Медицинские публикации
/
Авторы
/
Скрыпник А. В.
/
Розробка ПЛР тест-системи “TubMyc-Україна” для детекції ДНК бактерій роду Mycobacterium
УДК 619:579:616-076
Розробка ПЛР тест-системи “TubMyc-Україна” для детекції ДНК бактерій роду Mycobacterium
Стегній Б.Т., д-р. вет. наук, проф., акад. УААН,
Скрипник А.В., канд. вет. наук, с.н.с.
Національний науковий центр «Інститут експериментальної і клінічної ветеринарної медицини», м. Харків
У статті викладені результати випробувань специфічності ПЛР тест-системи, створеної в лабораторії молекулярної діагностики ІЕКВМ, для детекції бактерій роду Mycobacteriа. Також наведено дані секвенування видоспецифічних фрагментів гену, що кодує 16S рРНК, ізолятів мікобактерій, виділених в господарствах України, що дало можливість вперше отримати дані про циркуляцію деяких видів атипових мікобактерій на території України.
Приблизно третина населення земної кулі інфікована бактеріями, що належать до Mycobacterium tuberculosis complex. Переважна більшість інфікованих знаходиться в країнах, що розвиваються, де від туберкульозу вмирає 2,6 – 3 мільйона чоловік щорічно (Schütt-Gerowitt H., 1995; Rastogi N. et al., 2001).
Протягом останніх десятиріч атипові мікобактерії поступово відіграють все більшу роль як опортуністичні патогени, що можуть викликати захворювання (мікобактеріози) в імунодефіцитних організмах. У людей прикладом такої патології може бути дисемінована інфекція, зумовлена M. avium та M. genavense у хворих на СНІД. Серед тварин ураження імунної системи може бути викликано лейкозом великої рогатої худоби, який має значне поширення в Україні (Horsburgh C.R., 1991; Schütt-Gerowitt H., 1995; Horsburgh C.R. Jr., 1997; Бусол В.О. та ін., 2001).
В Україні протягом останніх десятиріч питома вага атипових мікобактерій, що виділяються лабораторіями ветеринарної медицини, зростає
високими темпами: від 15,1% (від загальної кількості виділених культур) в 1984 році (Завгородній А. І., 1997) до 67,9% в 2001 році (звітність Центральної лабораторії вет медицини, 2002).
Загальновідомим є той факт, що атипові мікобактерії обумовлюють позитивну алергічну реакцію на туберкулін. Згідно з „Інструкцією про заходи з профілактики та оздоровлення тваринництва від туберкульозу”, пукт 2.7.1, при виявленні реагуючих на туберкулін тварин в благополучних господарствах діагностичному забою піддають не менше 10 голів з кожного гурту. Згідно з пунктом 2.8 в неблагополучних господарствах усіх реагуючих на туберкулін тварин здають на забій.
Тому виникає необхідність швидкої та точної детекції мікобактерій для подальших діагностичних досліджень, зокрема ізолювання та диференціації. Незважаючи на наявність широкого спектру методів лабораторної діагностики цих збудників, їх детекція і ідентифікація залишається складною у зв’язку з повільними ростом на живильних середовищах мікобактерій багатьох видів, складністю їх виділення безпосередньо з біоматеріалу, виділення мікобактерій зі зміненою клітинною оболонкою, ультрадрібних та L-форм, недосконалістю існуючих методів диференціації (Нестеренко та ін., 1994).
Головним завданням діагностики є швидка та високо специфічна детекція мікобактерій в первинному клінічному та патологоанатомічному матеріалі. Це має велике значення з огляду на економію часу та коштів, затрачених на культивування. При проведенні моніторингових досліджень в господарствах та при проведенні патологоанатомічних досліджень не завжди можна виявити первинні осередки туберкульозних уражень.
Детекція і диференціація мікобактерій на основі ензиматичних тестів відносно проста й не вимагає значних витрат для її проведення. Однак вона досить тривала, вимагає наявності певної кількості бактеріальної маси, а для багатьох нових видів мікобактерій специфічні тести взагалі не розроблені. За кордоном, для виділення та ідентифікації мікобактерій сьогодні застосовуються молекулярні методи, що є більш досконалими в порівнянні з біохімічними тестами (Parish T., Stoker N.G., 1998).
Здатність мікобактерій існувати в деструктивних по клітинній оболонці та L-формах не дозволяє визначення за морфологічними ознаками класичних мікобактерій (трохи зігнуті червоні палички). Ці форми, на відміну від бактеріальної структури, не стійкі до дії кислот та алкоголю, що призводить до хибно-негативних результатів при фарбуванні мазків за методикою Циль-Нільсена.
Мікобактерії належать до підотряду Corynebacterineae, тому є генотипово та фенотипово споріднені до родів Nocardia, Rhodococcus, Corynebacterium, що також входять до цього таксону. Останні можуть в деяких випадках проявляти часткову кислотостійкість, внаслідок чого має місце часткове фарбування клітини у червоний колір за Циль-Нільсеном. Це може давати хибно-позитивні результати (Определитель бактерий Берджи, 1997; Dostal S. et al., 2003).
Полімеразна ланцюгова реакція (ПЛР) дозволяє виявляти ДНК мікобактерій в клінічному матеріалі навіть при наявності незначної кількості мікробних клітин, що є головною перевагою методу. Висока чутливість досягається в результаті ампліфікації (клонування) таргетної ділянки – концентрація специфічної олігонуклеотидної послідовності в реакційній пробі зростає в десятки мільйонів разів.
Полімеразна ланцюгова реакція дозволяє проводити оздоровчі заходи у коротший час, що дає змогу ефективно впливати на ситуацію в неблагополучних господарствах.
Метод секвенування дозволяє робити висновки щодо варіабельності мікобактерій, відкриваючи шлях до виділення нових підвидів і навіть видів, удосконалюючи тим самим систематику (Colin A. et al, 2002; Parish T. et al, 2001; Rastogi N. et al, 2001).
Для ідентифікації мікроорганізмів секвенуванням особливе значення мають гени 16S і 23S рибосомальної РНК. Вони знайдені в усіх організмах за винятком вірусів. На відміну від фенотипових та біохімічних властивостей видова послідовність 16S рРНК - стабільної природи та є специфічною для мікроорганізмів на видовому рівні. Для діагностичних цілей надзвичайно корисні гіперваріабельні ділянки в зв’язку з відносно великою різницею між видами і відносно низької варіабельності усередині виду (Rogall T. et al, 1990a,b; Bottger E.C., 1996).
Виходячи з цього метою наших досліджень було розробити ПЛР тест-сиcтему з праймерами, специфічними до ділянки гену 16S рибосомальної РНК для детекції мікобактерій.
Матеріали і методи. Дослідження проводили в лабораторії молекулярної діагностики ІЕКВМ УААН, лабораторії генної інженерії та національній референс-лабораторії вивчення туберкульозу і паратуберкульозу великої рогатої худоби Федерального дослідного інституту здоров’я тварин імені Ф. Льофлера, м. Йєна, Німеччина.
99 досліджуваних культур мікобактерій, виділених від ВРХ, свиней, собаки, з навколишнього середовища, сільськогосподарської, зоопаркової та синантропної птиці, було отримано з музею лабораторії вивчення туберкульозу ННЦ «ІЕКВМ», Одеської дослідної станції ННЦ «ІЕКВМ», Чернігівського інституту сільськогосподарської мікробіології, Київської міської, Луганської, Миколаївської, Херсонської та Харківської обласних державних лабораторій ветеринарної медицини.
В якості праймерів були використані олігонуклеотиди, комплементарні ділянці гену 16S рибосомальної РНК з 9 по 1046 позицію, відповідно до картування рРНК E.coli.
Програма реакції ампліфікації складалась з первинної денатурації при 96 °С протягом 1 хв, послідуючих 30 циклів: денатурація при 96 °С протягом 15 сек, відпал праймерів при 58 °С протягом 1 хв, елонгація праймерів при 72 °С протягом 1 хв (в останньому циклі час фінальної елонгації склав 5 хв).
При візуалізації продукту ампліфікації після електрофорезу його довжина складала 1037 пар нуклеотидів (п.н.).
В якості негативного контролю використовували воду, вільну від нуклеаз, в якості позитивного – штам DSM 44135 (M. a. paratuberculosis).
Для підтвердження результатів, отриманих за допомогою тест-ситеми, продукти ампліфікації секвенували. Для цього використовували набір «QIAquick Gel Extraction Kit» фірми QIAGEN.
Ампліфікацію ПЛР-продукту проводили в одному напрямку з одним праймером. Ділянка, що секвенували, була ампліфікована в ПЛР за наступною програмою: початкова денатурація при 96 °С протягом 1 хв та слідуючі 25 циклів: денатурація при 96 °С протягом 30 сек, відпал праймерів при 50 °С протягом 1 хв, елонгація праймерів при 60 °С протягом 4 хв.
Довжина ПЛР-продукту була у межах 450-500 п.н. і залежала від концентрації полімерази та часу синтезу.
Секвенування проводили за допомогою автоматичного флюоресцентного ДНК секвенатора ABI PRISM 310 Genetic Analyser (Applied Biosystems).
Підтвердження специфічності розробленої тест-системи проводили шляхом ампліфікації ДНК штамів Escherichia coli, Nocardia asteroides, Rhodococcus rhodochrous, Corynebacterium pseudotuberculosis
Результати досліджень. Гомологічність олігонуклеотидів різним видам мікобактерій визначали in silico за допомогою програми пошуку гомологічних послідовностей BLAST, з використанням баз даних GeneBank, EMBL (Європейська молекулярно-біологічна бібліотека), DDBJ (Японська база даних нуклеотидних послідовностей) та RIDOM (проект рибосомальної диференціації мікроорганізмів). В результаті перевірки в базах даних мережі Internet комплементарності до праймерів інших послідовностей ДНК бактерій, вірусів або еукаріот не виявлено.
Протокол реакції ампліфікації було оптимізовано: температуру відпалу праймерів підвищили до 58 °С, що підвищило специфічність; кількість циклів зменшили до 30, що дало можливість скоротити час реакції і водночас залишити її якість на високому рівні.
Внутрішньородову універсальність розробленої нами тест-системи “TubMyc-Україна” визначали шляхом дослідження 100 культур бактерій, що належать до 18 видів роду Mycobacterium, визначених за допомогою секвенування ділянки 16S рибосомальної РНК (Табл 1).
|
Кількість культур |
Вид мікобактерій, визначений за допомогою секвенування |
|
1 |
Mycobacterium frederiksbergense |
|
1 |
Mycobacterium engbackii |
|
29 |
Mycobacterium avium complex |
|
26 |
Mycobacterium fortuitum |
|
1 |
Mycobacterium doricum |
|
1 |
Mycobacterium parascrofulaсeum |
|
14 |
Mycobacterium phlei |
|
1 |
Mycobacterium thermoresistibile |
|
2 |
Mycobacterium duvalii |
|
1 |
Mycobacterium hassiacum |
|
16 |
Mycobacterium tuberculosis complex |
|
1 |
Mycobacterium smegmatis |
|
1 |
Mycobacterium paratuberculosis* |
|
1 |
Mycobacterium scrofulaceum |
|
1 |
Mycobacterium confluentis |
|
1 |
Mycobacterium madagaskariense |
|
1 |
Mycobacterium elephantis |
|
2 |
Mycobacterium nonchromogenicum |
Таблиця 1. Результати досліджень тест-системою “TubMyc-Україна”, та видова приналежність бактерій, визначена за допомогою секвенування (Dostal S. et al., 2003).
* M. paratuberculosis отримана з Німецького музею мікроорганізмів (DSM 44135)
В доступній літературі відомостей про циркуляцію, виділення або типування на території України таких видів мікобактерій, як M. frederiksbergensе, M. engbackii, M. doricum, M. hassiacum, M. confluentis, M. madagaskariense, M. elephantis ми не зустрічали.
На рисунку 1 представлені результати електрофорезу продуктів ампліфікації фрагменту гена, що кодує 16S рибосомальну РНК вище перерахованих видів мікобактерій. Дані рисунку переконливо свідчать про специфічність розробленої тест-системи, оскільки в усіх випадках продукт ампліфікації співпадав з позитивним контролем і був характерного розміру 1037 п.н.
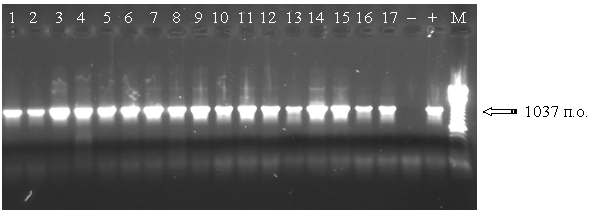
Рисунок 1. Результати електрофорезу продуктів ампліфікації: 1 – M. frederiksbergensе, 2 – M. engbackii, 3 – M. avium complex, 4 – M. fortuitum, 5 – M. doricum, 6 – M. parascrofulaсeum, 7 – M. phlei, 8 – M. thermoresistibile, 9 – M. duvalii, 10 – M. hassiacum, 11 – M. tuberculosis complex, 12 – M. smegmatis, 13 – M. nonchromogenicum, 14 – M. scrofulaceum, 15 – M. confluentis, 16 – M. madagaskariense, 17 – M. elephantis, ”–“ вода вільна від нуклеаз, “+” позитивний контроль M. paratuberculosis, M – маркер молекулярної ваги.
Контроль специфічності тест-ситеми проводили шляхом постановки ПЛР з ДНК 4 штамів бактерій, що не відносяться до роду Mycobacterium: Escherichia coli, Nocardia asteroides, Rhodococcus rhodochrous, Corynebacterium pseudotuberculosis. Результати цих досліджень у всіх випадках були негативними, що є характеристикою специфічності тест-системи.
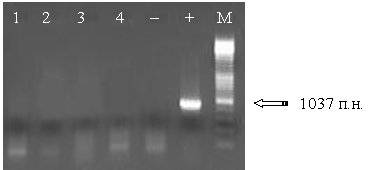
Рисунок 2. Результати ампліфікацї штамів: 1 – Escherichia coli, 2 – Nocardia asteroides, 3 – Rhodococcus rhodochrous, 4 – Corynebacterium pseudotuberculosis, ”–“ вода вільна від нуклеаз, “+” позитивний контроль M. paratuberculosis, M – маркер молекулярної ваги.
Висновки. Створена високоефективна тест-система “TubMyc-Україна” для виявлення мікобактерій різних видів. Праймери, що використовуються в даній тест-системі для детекції мікобактерій комплементарні тільки фрагменту гену, що кодує 16S рРНК мікобактерій.
Вперше було секвеновано видоспецифічні нуклеотидні послідовності фрагментів гену 16S рДНК в ізолятах мікобактерій, виділених на території України.
Вперше отримані дані про циркуляцію таких видів атипових мікобактерій, як M. frederiksbergensе, M. engbackii, M. doricum, M. hassiacum, M. confluentis, M. madagaskariense, M. elephantis на території України.
Список літератури:
1. Бусол В.О., Постой В.П., Коваленко А.М. Закономірності епізоотичного і епідемічного процесів при туберкульозі ВРХ та людей // Наукова конференція професорсько-викладацького складу, наукових співробітників та аспірантів за наслідками науково-дослідних робіт 2000 р.: Тези доп. – К., 2001. – С. 20.
2. Завгородний А.И. Виды микобактерий, распространенные в хозяйствах Украины, и их эпизоотическое значение. Дис… д-ра веет наук: 16.00.03. – Х., 1997. – 298с.
3. Інструкція про заходи з профілактики та оздоровлення тваринництва від туберкульозу // Тваринництво України №2. – 1994. – с.14-18.
4. Нестеренко Л.Н., Аксенов М.Ю., Гришина Т.Д., Шагинян И.А. и др. Тест-системы на основе полимеразной цепной реакции для идентификации возбудителя туберкулеза // Проблемы туберкулеза №2. – 1994. – с. 29-32.
5. Определитель бактерий Берджи / Под ред. Дж.Хоулта и др.; 9-е издание, в 2 т., т. 2. – М.: «Мир», 1997. – с. 607.
6. Bottger E.C. Approaches for identification of microorganisms // ASM News.- 1996, 62. - P.247-250.
7. Colin A. Graham, Alison J.M. Hill. DNA sequencing protocols - Totowa, New Jersey: Humana press, 2002.
8. Dostal S., Richter E., Harmsen D. Concise guide to mycobacteria and their molecular differentiation. - Würzburg, Germany: Ridom Press, 2003. – p.206.
9. Horsburgh C.R. Mycobacterium avium complex infections in the acquired immunodeficiency syndrome // N. Engl. J. Med. – 1991, 324. - P.1332-1338.
10. Horsburgh C.R. Jr. Epidemiology of Mycobacterium avium Complex Disease // American Journal of Medicine.- 1997, 102 (5C). – P.11-15.
11. Parish T., Stoker N.G. Mycobacteria protocols. - Totowa, New Jersey, USA: Humana Press, 1998.- p.472.
12. Parish T., Stoker N.G. Mycobacterium tuberculosis protocols. - Totowa, New Jersey, USA: Humana Press, 2001.
13. Rastogi N., Legrand E., Sola C. The mycobacteria: an introduction to nomenclature and pathogenesis // Rev. sci. tech. Off. Int. Epiz.- 2001, 20 (1), p.21-54.
14. Rogall T., Wolters J., Flohr T., Bottger E. Towards a phylogeny and definition of species at the molecular level within the genus Mycobacterium // International Journal of Systematic Bacteriology. – Oct. 1990, Vol. 40, No.4. - P.323-330.
15. Rogall T., Flohr T., Bottger E.C. Differentiation of mycobacterium species by direct sequencing of amplified DNA // J. Gen. Microbiol. – 1990, 136, р.1915-1920.
16. Schütt-Gerowitt H. On the development of Mycobacterial infections // Zbl. Bakt.- 1995, 283. - P.5-13.
“TubMyc-Україна” diagnostic kit development for the DNA detection of bacteria of genus Mycobacterium by polymerase chain reaction
Skrypnyk А.V., DVM, PhD, SR
Stеgniy B.Т., DVM, Prof., Acad. UAAS
National Scientific Center «Institute of Experimental and Clinical Veterinary Medicine», Kharkiv
In the paper the results of “TubMyc-Україна” diagnostic kit development and approbation for the bacterium DNA detection of genus Mycobacteria by polymerase chain reaction are described. Basing on the 16S rDNA gene partial sequence of Mycobacteria isolated in Ukrainian farms firstly reported data concerning the circulation of some atypical Mycobacteria species in the territory of Ukraine.
|



