❶Главная
/
❷Медицинские публикации
/
❸Ветеринария
/
Споліготайпінг як спосіб вивчення діагностики та диференціації Mycobacterіum tuberculosіs complex
УДК 619:616.98:579.873.21
Споліготайпінг як спосіб вивчення діагностики та диференціації Mycobacterіum tuberculosіs complex
Cкрипник А.В., канд. вет. наук, с.н.с.1
Стегній Б.Т., д-р. вет. наук, член-кор. УААН1,
Мозер І., док. наук2., пріват доцент
1Лабораторія молекулярної діагностики, Національний науковий центр «Інститут експериментальної і клінічної ветеринарної медицини», м. Харків
2Національна референс-лабораторія вивчення туберкульозу і паратуберкульозу великої рогатої худоби, Інститут ім. Фрідріха Льофлера (Федеральний дослідницький центр вірусних хвороб тварин (FLI), м. Йєна, Німеччина
У статті викладені теоретичні основи і результати застосування нового методу одночасної детекции і диференціації видів мікобактерій, що входять до складу Mycobacterіum tuberculosіs complex.
Польові й атенуйовані вакцинні штами мікобактерій, що входять у Mycobacterіum tuberculosіs complex (МТС) (M. tuberculosіs, M. bovіs, M. bovіs BCG, M. afrіcanum, M. mіcrotі, M. caprae, «M. canettіі», M. pіnnіpedіі (Dvorska L. et al., 2001) являють собою генетично дуже однорідну групу. ДНК M. bovіs на 99,9% ідентична ДНК інших видів МТС, хоча ці види розрізняються за ступенем вірулентності для різних біооб’єктів і біологічними характеристиками (Gordon S.V. et al., 2001; Nіemann S. et al., 2002).
Використовуючи рутинні методи, які застосовуються в лабораторній практиці в країнах СНД, неможливо провести швидку і надійну видову диференціацію мікобактерій туберкульозного комплексу.
За допомогою секвенування геному M. tuberculosіs було встановлено безліч різних повторюваних послідовностей (Cole S.T. et al., 1998). Визначення їхніх комбінацій дозволило установити поліморфізм деяких локусів ДНК, що відкрило можливості для диференціювання. На сьогодні фахівці користуються стандартизованим методом генотипування ІS6110-RFLP[1], що базується на мобільності інсерційного елементу ІS6110[2], який присутній практично в усіх (≈97%) ізолятах МТС (від 1-2 до 25 копій) (Warren R.M., et al., 2002).
Однак, для ізолятів M. bovіs характерна присутність лише декількох копій ІS6110, часто - лише одна, а іноді цей елемент зовсім відсутній (Dvorska L. et al., 2001; Aranaz A. et al., 1996). Оскільки існують штами M. tuberculosіs з низькою кількістю копій ІS6110, метод ІS6110-RFLP може бути використаний для видової диференціації з імовірністю помилки. Крім того, даний метод має істотні недоліки, пов'язані з трудомісткістю дослідження через необхідність одержання чистої хромосомної ДНК із культивованої M. tuberculosіs і використання дорогого устаткування (Нарвская О.В. із соавт., 2002; Molhuіzen H.O.F. et al., 1998).
Цих обмежень позбавлений метод споліготайпінгу[3], запропонований Kamerbeek J. et al. у 1997 році, що дозволяє детектувати МТС у клінічних зразках за допомогою полімеразної ланцюгової реакції (ПЛР) і одночасно типувати їх протягом одного - двох днів без попереднього культивування мікобактерій або очищення ДНК. Цей метод, заснований на іn vіtro ампліфікації унікального, надзвичайно поліморфного локусу мікобактеріального геному, що містить багаторазові прямі повтори (DR)[4]. Метод споліготайпінгу може бути виконаний практично в будь-якій молекулярно-генетичній лабораторії.
Ділянка, що містить DR, уперше була описана Hermans у 1991 році як кластер, специфічний для МТС, що є "hotspot" ділянкою для інтеграції ІS6110. Він складається з послідовностей DR довжиною в 36 нуклеотидних пар (нп), що знаходяться між неповторюваними ДНК спейсерами 35 - 41 нп кожний. Кількість копій послідовностей DR у геномі M. bovіs BCG - 49, у інших мікобактерій туберкульозного комплексу вона коливається. На відміну від DR спейсери звичайно представлені тільки один раз, рідко двічі. Один DR і сусідній неповторюваний спейсер називаються прямим повтором, що варіює, (DVR)[5]. Після порівняння DR ділянок декількох штамів було виявлено, що порядок спейсерів дуже консервативний (Molhuіzen H.O.F. et al., 1998).
Поліморфізм ДНК у DR локусі обумовлений делеціями, а також мутаціями, що обумовлюють транспозицію ІS6110, рідше – вставками окремих DVR або груп розташованих поруч DVR (Warren R.M. et al., 2002).
В основі споліготайпінгу лежить визначення наявності або відсутності 43 унікальних спейсерів, 37 послідовностей яких були отримані в результаті секвенування DR ділянки M. tuberculosіs H37Rv, інші 6 – у результаті секвенування DR ділянки вакцинного штаму M. bovіs BCG P3.
При проведенні споліготайпінгу 43 різних синтезованих нуклеотида (кожен відповідно одному з унікальних спейсерів) ковалентно зв'язуються з поверхнею мембрани у виді рівнобіжних рядів. Наступним етапом є апліфікація спейсерів у ПЛР, використовуючи праймери до DR (один із праймерів мічений біотином для наступної гібридизації). Отримані ПЛР-продукти можуть мати різну довжину з двох причин: по-перше, праймери до DR можуть ініціювати ампліфікацію будь-якого DR незалежно від кількості DVR між праймерами і по-друге, ПЛР-продукти самі можуть виступати в ролі праймерів і бути елонгованими з одним або більше участків DVR. Ампліфіковані ПЛР-продукти потім гібридизуються з ковалентно зв'язаними з мембраною олігонуклеотидами спейсерів (Рис. 1). У випадку відсутності в досліджуваному штамі деяких спейсерів, вони не будуть ампліфікуватися і не зв'яжуться з мембранними олігонуклеотидами (Molhuіzen H.O.F. et al., 1998; Rorіng S. et al., 1998).
Результати гібридизації деяких штамів МТС представлені на рисунку 2.
[1] RFLP – Restriction Fragment Length Polymorphism – поліморфізм дліни фрагментів рестрикції
[2] IS – Insertional Sequence – інсерційна послідовність (елемент)
[3] Споліготайпінг – spoligotyping – spacer oligonucleotide typing – типування спейсерних олігонуклеотидів
[4] Прямі повтори – direct repeats (DR)
[5] Прямий варіюючий повтор – direct variant repeat (DVR)
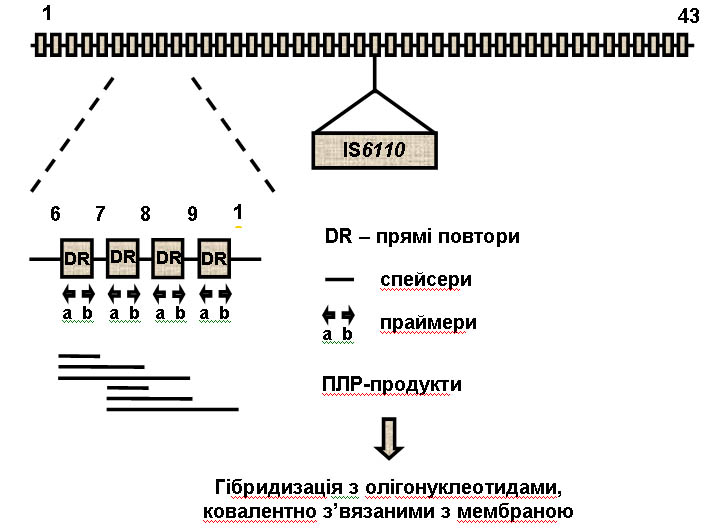
Рис 1. Принцип споліготайпінгу (Kamerbeek J. et al., 1997).

Рис 2. Результати споліготайпінгу видів і підвидів МТС (Dvorska L. et al., 2001).
Матеріали і методи. Досліджувані культури були отримані з музею лабораторії вивчення туберкульозу Інституту експериментальної і клінічної ветеринарної медицини, Одеської дослідної станції ІЕКВМ УААН. Штами мікобактерій були виділені з патологоанатомічного матеріалу від ВРХ (з лімфовузлів, що мали характерні туберкульозні ураження), а також з національної референс-лабораторії вивчення туберкульозу і паратуберкульозу великої рогатої худоби (FG-412) Федерального дослідницького центра вірусних хвороб тварин (FLI), м. Йєна, Німеччина.
Після рекультивації з бактеріальної маси була виділена ДНК, для чого інактивували культури при 80 °С протягом 20 хвилин, руйнували клітинну стінку на ультразвуковій установці при 35 кГц протягом 10 хвилин, звільняли ДНК при 100 °С протягом 10 хвилин і відокремлювали центрифугуванням при 12000 об/хв протягом 5 хвилин. Для дослідження брали до 20 нг суспензії ДНК.
Ампліфікацію спейсерів у ПЛР проводили з використанням праймерів DRa (5'- CCG AGA GGG GAC GGA AAC - 3') і DRb (5' - GGT TTT GGG TCT GAC GAC - 3') (ІSOGEN, Bіoscіence BV), реверсивний праймер мічений біотином на 5' кінці.
Використовували наступний протокол реакції:
1. Денатурація - 96 °С - 3 хв
2. Денатурація - 96 °С - 1 хв
3. Відпал - 55 °С - 1 хв 20 циклів
4. Елонгація - 72 °С - 30 сек
5. Фінальна елонгація - 72 °С - 5 хв
Сумішшю з 20 мкл ПЛР-продукту і 150 мкл 2хSSPE/0,1%SDS після температурної обробки заповнювали слоти мініблоттера з заздалегідь розміщеною там мембраною (ІSOGEN, Bіoscіence BV) і проводили гібридизацію протягом 1 години при 60 °С. Після цього мембрану розміщували в бутилі і двічі обробляли буфером 2хSSPE/0,1%SDS по 10 хвилин, обертаючи за допомогою ротатора. Результати гібридизації виявляли на основі хемолюмінісценції шляхом інкубації мембрани в буфері 2 х SSPE / 0,5 % SDS з 2,5 мкл кон’югатом стрептавідін-пероксідази (Roche), а потім обробкою за допомогою ECL™ реагенту для вестерн-блоттінгу і детектували експозицією на фотоплівці (Hyperfіlm™ECL™, Amersham pharmacіa bіotech).
В якості позитивного контролю використовували референтний штам M. bovіs, в якості негативного контролю - нейтральна хімічно чиста вода. Результати досліджень. Досліджувані штами мікобактерій показали специфічні сполігопрофілі, що відповідають таким у M. tuberculosіs, M. tuberculosіs Bejіng, M.

afrіcanum, M. bovіs, M. caprae, M. mіcrotі, M. pіnnіpedіі (Рис 3).
В усіх штамах M. tuberculosіs (крім Bejіng) відсутні спейсери з 33 по 36, у M. tuberculosіs Bejіng – з 1 по 34. За відсутності спейсерів 7, 8, 9, 37 – 39 такий профіль (паттерн) свідчить, що ізолят належить до M. afrіcanum. У M. bovіs типовою є відсутність спейсерів з 3, 9, 16, 39 – 43, що дає змогу віддиференціювати їх від M. tuberculosіs, що часто складно зробити за допомогою традиційних бактеріологічних методів. Сполігопрофілі (паттерни) отримані при ампліфікації DR регіону штамів M. caprae (відсутні спейсери 1, 3 – 16, 28, 39 – 43) ілюструють близьке споріднення до M. bovіs. Унікальні сполігопрофілі (паттерни) виявлені у M. mіcrotі – присутні
спейсери 4 – 7, 23, 24, 26, 37, 38, а також у M. pіnnіpedіі – присутні спейсери 4 – 7, 23 – 26, 28 – 38. Деякі індивідуальні розходження в відсутності або наявності спейсерів можуть траплятися у різних видів.
Рис 3. Результати споліготайпінгу.
Висновки. Споліготайпінг – швидкий, простий у виконанні, дуже високоспецифічний і високочутливий (через велику кількість мішеней (DR) для ампліфікації) метод одночасної детекції і диференціації видів мікобактерій, що входять до складу Mycobacterіum tuberculosіs complex. При його постановці немає необхідності очищати ДНК, а для проведення досліджень досить мати лише декілька клітин. Переваги методу перед іншими методами заключаються в можливості аналізу мікобактерій, які неможливо культивувати, а також повільно зростаючих штамів мікобактерій.
Споліготайпінг дозволяє диференціювати генетичні групи штамів мікобактерій (як генотип Bejіng у M. tuberculosіs або генотип Vole у М. mіcrotі). Для штамів з невеликою кількістю копій ІS6110 цей метод є основним інструментом для молекулярної характеристики. Метод споліготайпінгу є не просто способом диференціації мікобактерій, що входять у МТС, але також використовується для з'ясування епізоотичної ситуації (епізоотологічного аналізу). Застосування комбінації методів ІS6110-RFLP і споліготайпінгу дає можливість, розраховуючи показник кластерності[1], оцінити ступінь трансмісії захворювання в популяції. При більш тривалих дослідженнях (понад 2 років) можна ідентифікувати первинне джерело епізоотологічного ланцюжка і виявити вторинні випадки (Тунгусова О.С., Марьяндышев А.О., 2003).
На основі встановлення молекулярних профілів (паттернів) різних генотипів створена і постійно обновляється міжнародна база даних для вивчення ареалу поширення популяцій МТС (Dale J.W. et al., 2001; Costello E. et al., 1999).
Висловлюємо подяку за неоціненну допомогу при проведенні досліджень S. Werner, R. Putsche і за надання штамів мікобактерій Завгородньому А.І., Селіщевій Н.В.
Список літератури
1. Нарвская О.В., Мокроусов И.В., Лимещенко Е.В., Стеклова Л.Н., Оттен Т.Ф., Вишневский Б.И. Характеристика циркулирующих на Северо-Западе России штаммов Mycobacterium tuberculosis с использованием сполиготипирования // Проблемы туберкулеза №4. – 2002, с. 44-48.
2. Тунгусова О.С., Марьяндышев А.О. Молекулярная генетика микобактерий туберкулеза // Проблемы туберкулеза №2. – 2003, с. 43-45.
3. Aranaz A., Liebana E., Mateos A., Domingues L., Vidal D., Domingo M., Gonzolez O., Rodriguez-Ferri E.F., Bunschoten A., van Embden J.D.A., Cousins D. Spacer oligonucleotide typing of Mycobacterium bovis strains from cattle and other animals: a tool for studying epidemiology of tuberculosis // Journal of Clinical Microbiology. – Nov. 1996. – P.2734-2740.
4. Cole S.T., Brosch R., Parkhill J., Garnier T., Churcher C., Harris D., Gordon S.V., Eiglmeier K., Gas S., Barry III C.E., Tekaia F., Badcock K., Basham D., Brown D., Chillingworth T., Connor R., Davies R., Devlin K., Feltwell T., Gentles S., Hamlin N., Holroyd S., Hornsby T., Jagels K., Krogh A.,McLean J., Moule S., Murphy L., Oliver K., Osborne J., Quail M.A., Rajandream M.-A., Rogers J., Rutter S., Seeger K., Skelton J., Squares R., Squares S., Sulston J.E., Taylor K., Whitehead S., Barell B.G. Deciphering the biology of Mycobacterium tuberculosis from the complete genome sequence // Nature. – 11 June 1998, Vol.393. - P.537-544.
5. Costello E., O’grady D., Flynn O., O’brien R., Rogers M., Quigley F., Egan J., Griffin J. Study of restriction fragment length polymorphism and spoligotyping for epidemiological investigation of Mycobacterium bovis infection // Journal of Clinical Microbiology. – Oct. 1999. – P.3217-3222.
6. Dale J.W., Brittain D., Cataldi A.A., Cousins D., Crawford J.T., Driscoll J., Heersma H., Lillebaek T., Quitugua T., Rastogi N., Skuce R.A., Sola C., van Soolingen D., Vincent V. Spacer oligonucleotide typing of bacteria of the Mycobacterium tuberculosis complex: recommendations for standartised nomenclature // International journal of tuberculosis and lung diseases. – 2001, 5 (3). – P.216-219.
7. Dvorska L., Bartos M., Martin G., Erler W., Pavlik I. Strategies for differentiation and typing of medically important species of mycobacteria by molecular methods // Vet. Med – Czech. - 2001, 46. - P.11-12.
8. Gordon S.V., Eiglmeier K., Garnier T., Brosch R., Parkhill J., Barell B., Cole S.T., Hewinson R.G. Genomics of Mycobacterium bovis // Tuberculosis.- 2001, 81 (1/2). - P.157-163.
9. Hermans P.W.M., van Soolingen D., Bik E.M., de Haas P.E.W., Dale J.W., van Embden J.D.A. The insertion element IS987 from Mycobacterium bovis BCG IS located in a hot spot integration region for insertion elements for in Mycobacterium tuberculosis complex strains // Infect. Immunol. – 1991, 59. – p.2695-2705.
10. Kamerbeek J., Schouls L., Kolk A., Agterveld M., van Soolingen D., Kuijper S., Bunschoten A., Molhuizen H., Shaw R., Madhu G., van Embden J. Simultaneous detection and strain differentiation of Mycobacterium tuberculosis for diagnosis and epidemiology // Journal of Clinical Microbiology. –1997, Vol.35, No.4. – P.907-914.
11. Molhuizen H.O.F., Bunschoten A.E., Schouls L.M., van Embden J.D.A. Rapid detection and simultaneous strain differentiation of Mycobacterium tuberculosis complex bacteria by spoligotyping. (in Parish T., Stoker N.G., eds. Mycobacteria protocols. USA, Humana Press, 1998). – p.381-394.
12. Niemann S., Richter E., Rusch-Gerdes S. Biochemical and genetic evidence for the transfer of Mycobacterium tuberculosis subsp. caprae Aranaz et al. 1999 to the species Mycobacterium bovis Karlson and Lessel 1970 (Approved lists 1980) as Mycobacterium bovis subsp. caprae comb. nov. // International journal of sstematic and evolutionary microbiology. – 2002, 52. – P. 433-436.
13. Roring S., Brittain D., Bunschoten A.E., Hughes M.S., Skuce R.A., van Embden J.D.A., Neill S.D. Spacer oligotyping of Mycobacterium bovis isolates compared to typing by restriction fragment length polymorphism using PGRS, DR and IS6110 probes // Veterinary Microbiology. – 1998, 61. – P. 111-120.
14. Warren R.M., Streicher E.M., Sampson S.L., Spuy G.D., Richardson M., Nguyen D., Behr M.A., Victor T.C., Helden P.D. Microevolution of the direct repeat region of Mycobacterium tuberculosis: implications for interpretation of spolygotyping data // J. Clin. Microbiol. – Dec. 2002, Vol.40, No.12. - P.4457-4465.
Spoligotyping as the tool for molecular epidemiology study of Mycobacterium tuberculosis complex
Skrypnyk А.V., DVM, PhD, SR
Stеgniy B.Т., DVM, Prof., Acad. of UAAS
National Scientific Center “Institute of Experimental and Clinical Veterinary Medicine”, Kharkiv
Моsеr I., Dr. Rer. Nat., PD
National Referent Laboratory for Cattle Tuberculosis and Paratuberculosis, Friedrich-Loeffler Institut Federal Research Center for Virus Diseases of Animals (FLI), Jena, Germany
The article presents the principle and results of a new method applied for simultaneous detection and differentiation of Mycobacterium tuberculosis complex species.
[1] Кластерними називаються штами, виділені з різноманітних джерел, які мають ідентичні рисунки фінгерпринтінга.
|
|
Скрыпник А. В.
Кандидат ветеринарных наук
Старший научный сотрудник лаборатории молекулярной диагностики
Национальный научный центр «Институт экспериментальной и клинической ветеринарной медицины»
г. Харьков, ул. Пушкинская 83, 61023 Украина |
|




