❶Главная
/
❷Медицинские публикации
/
❸Ветеринария
/
Визначення ефективності in-house методів екстракції ДНК з бактеріальної маси мікобактерій
Визначення ефективності in-house методів екстракції ДНК з бактеріальної маси мікобактерій
Cкрипник А. В., канд. вет. наук, с.н.с.
Національний науковий центр «Інститут експериментальної і клінічної ветеринарної медицини», м. Харків
У статті викладено результати порівняльного аналізу п’яти in-house методів виділення ДНК з мікобактерій для подальшого її використання в полімеразній ланцюговій реакції з огляду на такі критерії, як: ефективність, тривалість виконання, трудомісткість, безпечність для здоров’я, витрати на реактиви і устаткування тощо. Показано, що найбільш відповідає цим критеріям розроблений нами спосіб обробки мікобактеріальних клітин високою температурою.
Туберкульоз – це небезпечна зооантропонозна інфекція, що становить загрозу епідеміологічному та економічному благополуччю. Збитки, яких завдає Mycobacterium bovis сільському господарству у світі, щороку сягають 3 млрд. доларів США [1]. Кожного року в світі від туберкульозу вмирає близько 2 млн. чоловік. В Україні це зооантропонозне захворювання також залишається надзвичайно гострою і актуальною проблемою як для гуманної, так і для ветеринарної медицини. Незважаючи на значні досягнення ветеринарної служби, захворювання тварин на туберкульоз реєструється в регіонах усіх природно-географічних зон і завдає суттєвих збитків тваринництву нашої держави [2, 3].
Результати лабораторних досліджень з урахуванням епізоотологічних даних є регламентуючими у постановці діагнозу на туберкульоз. У державних лабораторіях ветеринарної медицини України застосовують методи бактеріологічної діагностики, зокрема визначення фенотипових ознак мікобактерій (кислотостійкість, характер і швидкість росту, наявність або відсутність певних біохімічних характеристик, вірулентність для лабораторних тварин тощо) [4, 5, 6]. На часі стоїть впровадження у вітчизняну лабораторну практику сучасних молекулярно-генетичних методів діагностики туберкульозу, зокрема полімеразної ланцюгової реакції (ПЛР).
Суттєвим етапом застосування будь-якого молекулярно-генетичного методу досліджень є екстракція нуклеїнових кислот. Виділення нативних ДНК та РНК є ключовим моментом забезпечення високої якості результатів. Екстракція ушкоджених нуклеїнових кислот або виділення їх разом з інгібіторами полімерази може спричинити помітне зниження чутливості або взагалі хибно негативні результати [7].
Унікальна будова клітинної оболонки мікобактерій є перешкодою, яку важко здолати для одержання ДНК [8]. Тому стандартні методи її екстракції, розроблені для грам-негативних і грам-позитивних бактерій, не підходять для мікобактерій [9].
Для виділення геномної та плазмідної ДНК з мікобактерій розроблено багато різноманітних способів: хімічні, механічні, виділення за допомогою ферментів тощо. Використання тих або інших методик залежить від мети подальшого використання ДНК.
Класичними прикладами хімічного способу є фенол-хлороформна екстракція [10] та обробка мікобактеріальних клітин гуанідин-гідрохлоридом [11].
За допомогою механічних методів здійснюється фізичне руйнування клітинної оболонки мікобактерій. При цьому використовують, наприклад, скляні або цирконієві бусинки [12, 13]. До механічних методів можна також віднести раптову декомпресію клітин азотом [14] і руйнування клітин мікобактерій за допомогою сухого льоду [15]. Доволі простим у виконанні та швидким є метод обробки суспензії мікобактеріальних клітин ультразвуком [16]. Хоча ці методи ефективні для лізису клітин, їхніми хибами є руйнування геномної ДНК, потреба в спеціалізованому обладнанні та підвищена небезпечність створення аерозолів патогенних мікобактерій [9].
До методів екстракції ДНК за допомогою ферментів можна віднести обробку протеїназою К, ліпазою, лізоцимом або їх комбінацію [17, 18]. Для одержання необхідної кількості ДНК з використанням лізоциму необхідно спочатку здійснити органічну екстракцію або ферментативну руйнацію ліпофільних компонентів клітинної оболонки, щоб дати можливість лізоциму впливати на пептидоглікани [9].
Метою нашої роботи було дослідження ефективності найбільш розповсюджених та, водночас, простих in-house методів екстракції ДНК з бактеріальної маси мікобактерій для її подальшого використання в ПЛР; вдосконалити або розробити новий, більш ефективний з точки зору економічних затрат, трудомісткості та швидкості метод екстракції ДНК з біомаси мікобактерій.
Матеріали і методи. Дослід здійснювали з чотирма культурами атипових мікобактерій: M. smegmatis, M. avium subsp. avium, M. avium subsp. hominissuis, M. avium subsp. paratuberculosis, а також з двома референтними культурами, що входять до M. tuberculosis complex: M. caprae та M. bovis.
З метою оцінки ефективності виділення ДНК з бактеріальної маси мікобактерій різними методами суспензії мікобактерій було приведено до єдиного стандарту визначенням співвідношення їх оптичної щільності за довжини хвиль А260-А280 методом спектрофотометрії. Для стандартизації концентрації бактеріальних клітин суспензії розводили до значення найменшої отриманої оптичної щільності. Для екстракції ДНК з мікобактерій відбирали по 200 мкл стандартизованих таким чином суспензій для кожного методу.
Екстракція мікобактеріальної ДНК способом ультразвукової дезінтеграції клітин з подальшою їх обробкою високою температурою. Суспензії мікобактерій обробляли ультразвуком упродовж 10 хвилин у водяній бані за кімнатної температури. Після цього пробірки розміщували на водяній бані за температури 99–100 °С на 10 хвилин для вивільнення ДНК. Останню відділяли центрифугуванням при 12000 об/хв упродовж 5 хвилин. Одержаний супернатант використовували для проведення ПЛР досліджень.
Екстракція мікобактеріальної ДНК способом обробки клітин високою температурою. Суспензії мікобактерій витримували на водяній бані впродовж 10 хвилин за температури 99–100 °С. Клітинний детрит видаляли центрифугуванням при 12000 об/хв протягом 5 хвилин. Супернатант використовували для подальших досліджень в ПЛР.
Екстракція мікобактеріальної ДНК за допомогою впливу на клітини мікрохвильового випромінювання. Суспензії мікобактерій піддавали впливу мікрохвильового випромінювання потужністю 180 Вт упродовж 20 хвилин, після чого їх центрифугували при 12000 об/хв протягом 5 хвилин. Супернатант, що містив ДНК, використовували в ПЛР.
Екстракція мікобактеріальної ДНК способом руйнації клітин скляними бусинками. До суспензій мікобактерій додавали скляні бусинки та руйнували клітинну оболонку вихровим перемішуванням на вортексі. ДНК звільнювали від детриту бактерійних клітин центрифугуванням при 12000 об/хв упродовж 5 хвилин, супернатант використовували для досліджень у ПЛР.
Виділення мікобактеріальної ДНК способом фенол-хлороформної екстракції проводили, як описано в [10].
Для визначення ступеня ефективності різних методів виділення ДНК з мікобактерій для подальшої постановки ПЛР з кожного зразка ДНК робили десятикратні розведення до 103–104 та проводили ПЛР з родоспецифічною системою праймерів genusMyc F / genusMyc R [19].
Результати досліджень. Результати приведення суспензій бактеріальної маси мікобактерій до єдиного стандарту подані в таблиці 1.
Кожну зі стандартизованих таким чином суспензій використовували для досліджень з визначення ефективності екстракції мікобактеріальної ДНК для використання в ПЛР, яку проводили з десятикратними розведеннями зразків ДНК культур, виділеними в різний спосіб (рис 1).
Клітини M. а. paratuberculosis мають винятково товстий шар гліколіпідів, які являють собою значну перешкоду ефективному виділенню ДНК. Тому залучення M. а. paratuberculosis до досліду надало можливість здійснити максимально об’єктивну оцінку ефективності екстракції ДНК з мікобактерій (рис 1).
У результаті ампліфікації було одержано специфічний продукт (розміром приблизно 1000 п.н.) в розведеннях суспензій до 104 включно, отриманих усіма способами виділення ДНК за виключенням фенол-хлороформної екстракції. Розведення суспензії, здобутої останнім способом, були ампліфіковані тільки до 102 включно: в розведеннях 103 та 104 специфічного продукту виявлено не було (рис. 1).
Порівняння часу проведення екстракції ДНК різними методами наведене в таблиці 2. Найшвидшим і нетрудомістким виявився метод руйнування клітинної оболонки скляними бусинками: результат може бути отриманий за 6 хвилин. Найдовшим і найбільш трудомістким виявився метод фенол-хлороформної екстракції, процес одержання результату цим методом займає близько 2,5 годин.
Інші методи екстракції ДНК виявились приблизно однаковими з огляду на затрати часу та трудомісткість.
Обговорення результатів. При дослідженні in-house методів екстракції ДНК з бактеріальної маси мікобактерій для її подальшого використання в ПЛР з огляду на такі критерії, як ефективність, швидкість та зменшення затрат матеріальних і людських ресурсів, було здійснено порівняльний аналіз п’яти методів виділення ДНК.
Найгірші результати були отримані під час застосування класичного методу фенол-хлороформної екстракції. Цей метод виявився найменш ефективним, застосовуючи його необхідно мати справу з небезпечними для здоров’я хімікатами, здійснювати багато маніпуляцій, тривалість виділення ДНК становить приблизно 2,5 години. Хоча за допомогою цього способу й можна отримати ДНК дуже високої якості, проте для використання екстрактів у ПЛР це має другорядне значення.
Порівняно з ним методи фізичної екстракції набагато швидші – від 6 хвилин (руйнація клітин скляними бусинками) до 25 хвилин (решта методів). Однак, усі вони вимагають як наявності відповідного устаткування (водяних бань, ультразвукових дезінтеграторів тощо), так і необхідності придбання реагентів.
За рівної ефективності виділення ДНК найпростішим і найдешевшим методом виявився запропонований нами метод екстракції мікобактеріальної ДНК шляхом обробки клітин високою температурою 99–100 °С протягом 20 хвилин.
Висновки. З огляду на такі критерії, як швидкість, трудомісткість, витрати на реактиви та обладнання, найбільш ефективним, на нашу думку, є запропонований нами спосіб обробки мікобактеріальних клітин високою температурою (Деклараційний патент України № 8094 від 15.07.2005). Цей спосіб не поступається за ефективністю іншим лабораторним методам, дозволяє здійснити екстракцію ДНК протягом 20 хвилин і не потребує реагентів.
Перспективи. Запропонований нами метод екстракції ДНК з бактеріальної маси мікобактерій може бути застосований в діагностичних та дослідницьких лабораторіях, що працюють в області ветеринарної та гуманної фтизіатрії.
Список літератури
1. The complete genome sequence of Mycobacterium bovis / T. Garnier [et al.] // PNAS. – 2003. – Vol. 100, №.13. – P.7877-7882.
2. До питання діагностики туберкульозу тварин / Ю. Колос [та ін.] // Ветеринарна медицина України. – 2006. – № 11. – С.10-12.
3. Ткаченко, О.А. Туберкульоз і мікобактеріозна інфекція великої рогатої худоби: автореф. дисс… доктора вет. наук. – Київ, 1999. – 36 с.
4. Дяченко, Г. Проблема діагностики туберкульозу сільськогосподарських тварин у сучасних умовах / Г. Дяченко, Н. Кравченко, В. Романенко // Ветеринарна медицина України. – 2006. – № 1. – С.5-7.
5. Завгородний, А.И. Виды микобактерий, распространенные в хозяйствах Украины, и их эпизоотическое значение: дис… д-ра вет наук: 16.00.03 / Завгородний Андрей Иванович. – Х., 1997. – 298с.
6. Методические рекомендации по уточнению диагноза на туберкулез у крупного рогатого скота благополучных хозяйств и определению видовой принадлежности культур микобактерий / Ю.Я. Кассич [и др.] // Украинский научно-исследовательский институт экспериментальной ветеринарии. – Х., 1987. – 19 с.
7. Bartlett, J.M.S. Extraction of nucleic acid templates / J.M.S. Bartlett, D. Stirling // PCR protocols, 2nd ed. Methods in molecular biology. – Totowa: Humana Press, 2003. – Vol. 226. – P.27-29.
8. Brennan, P.J. The envelope of Mycobacteria / P.J. Brennan, H. Nikaido // Annu. Rev. Biochem. – 1995. – Vol. 64. – P.29-63.
9. Belisle, J.T. Isolation of genomic DNA from Mycobacteria / J.T. Belisle, M.G. Sonnenberg // Methods in molecular biology / Eds. T. Parish, N.G. Stoker. – Totowa: Humana Press, 1998. – Vol. 101. – P.323-347.
10. Sambrook, J. Molecular Cloning: A Laboratory Manual / J. Sambrook, F. Fritsch, T. Maaniatis. – 2nd ed. – New York: Cold Spring Harbor, 1989.
11. A novel method for the isolation of mycobacterial DNA / J.A. Gongalez-y-Merchand [et al] // FEMS Microbiol. Lett. – 1996. – Vol.135. – P.71-77.
12. Barrera, L.F. Assessment of mycobacterial infection and multiplication in macrophages by polymerase chain reaction / L.F. Barrera, E. Skamene, D. Radzioch // Journal of Immunological Methods. – 1993. – Vol. 157. – P.91-99.
13. Genetic systems for mycobacteria / W.R. Jacobs [et al.] // Methods Enzymol. – 1991. – Vol. 204. – P.537-555.
14. Yandell, P.M. Isolation of deoxyribonucleic acid from Mycobacterium avium by rapid nitrogen decompression / P.M. Yandell, C. McCarthy // Infect. Immun – 1980. – Vol. 27. – P.368-375.
15. Preparation of high-molecular weight DNA: application to mycobacterial cells / T. Imai [et al.] // Anal. Biochem. – 1994. – Vol. 222. – P.479-482.
16. PCR-based detection of mycobacteria in sputum samples using a simple and reliable DNA extraction protocol / U. Reischl [et al.] // Bio Techniques. – 1994. – Vol. 17. – P.844-845.
17. Isolation and analysis of restriction endonuclease digestive patterns of chromosomal DNA from Mycobacterium paratuberculosis and other Mycobacterium species / D.L. Whipple [et al.] // Journal of Clinical Microbiology. – 1987. – Vol. 25. – P.1511-1515.
18. Mizuguchi, Y. Method for isolation of deoxyribonucleic acid from mycobacteria / Y. Mizuguchi, T. Tokunaga // Journal of Bacteriology. – 1970. – Vol. 104. – P.1020-1021.
19. Коваленко, А. М. Розробка ПЛР тест-системи «TubMyc-Україна» для детекції бактерій роду Mycobacterium / А. М. Коваленко, Б. Т. Стегній, А. В. Скрипник // Ветеринарна медицина: Міжвід. темат. наук. зб. – Х., 2005. – Вип. 85. – С. 528–533
Efficiency determination of in-house methods of DNA extraction from mycobacterial bacterial mass
Skrypnyk А., DVM, PhD, SR
National Scientific Center «Institute of Experimental and Clinical Veterinary Medicine»,
Kharkiv
Abstract
The article presents the results of comparative analysis of five in-house methods of DNA extraction from Mycobacteria to use in polymerase chain reaction subjected to such criteria: effectiveness, performance duration, labour-intensiveness, health safety, costs for reagents and equipment. It is shown that the most applicable is method of mycobacterial cell processing with high temperature.
Таблиця 1 – Результати вимірювання співвідношення оптичної щільності суспензій культур мікобактерій за співвідношення довжини хвилі А260-А280 та обчислення ступеня розведення для виготовлення стандартних суспензій
|
Культура |
Співвідношення оптичної щільності за А260-А280 |
Розведення |
|
M. smegmatis |
1,648 |
1 : 22,6 |
|
M. smegmatis |
0,75 |
1 : 10,3 |
|
M. smegmatis |
0,83 |
1 : 11,4 |
|
M. smegmatis |
0,42 |
1 : 5,8 |
|
M. smegmatis 1 |
0,294 |
1 : 4 |
|
M. smegmatis 2 |
0,451 |
1 : 6,2 |
|
M. smegmatis 4 |
0,431 |
1 : 5,9 |
|
M. a. avium |
0,087 |
1 : 1,2 |
|
M. a. avium 1 |
0,38 |
1 : 5,2 |
|
M. a. avium 2 |
0,073 |
1 : 1 |
|
M. a. avium 3 |
0,387 |
1 : 5,3 |
|
M. a. hominisuis |
0,182 |
1 : 2,5 |
|
M. a. hominisuis 1 |
0,427 |
1 : 5,8 |
|
M. a. hominisuis 2 |
0,275 |
1 : 3,8 |
|
M. a. hominisuis 3 |
0,248 |
1 : 3,4 |
|
M. a. paratuberculosis 1 |
0,182 |
1 : 2,5 |
|
M. a. paratuberculosis 2 |
0,113 |
1 : 1,5 |
|
M. a. paratuberculosis 3 |
0,42 |
1 : 5,8 |
|
M. a. paratuberculosis 4 |
0,129 |
1 : 1,8 |
|
M. caprae 2729 |
0,282 |
1 : 3,9 |
|
M. bovis 1119 |
0,352 |
1 : 4,8 |
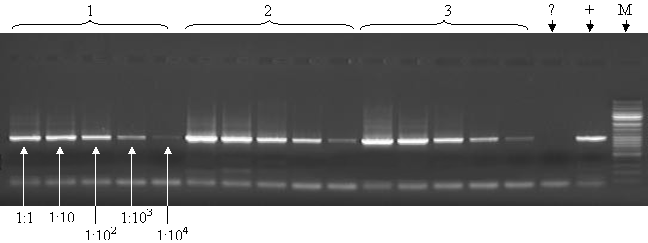

Рис. 1 Електрофореграма результатів проведення ПЛР з ДНК Mycobacterium avium subsp. paratuberculosis, екстрагованої різними методами
1 – екстракція мікобактеріальної ДНК способом ультразвукової дезінтеграції клітин з подальшим їх нагріванням за високої температури; 2 – екстракція ДНК способом нагрівання клітин за високої температури (99–100 °С); 3 – екстракція ДНК за допомогою впливу на клітини мікрохвильового випромінювання; 4 – екстракція ДНК способом руйнації клітин скляними бусинками; 5 – екстракція ДНК способом фенол-хлороформної екстракції; «−» – негативний контроль; «+» – позитивний контроль; «М» – маркер молекулярної ваги; «1:1 - 1:104» – десятикратні розведення зразків ДНК.
Таблиця 2 – Тривалість проведення екстракції ДНК різними методами
|
Метод екстракції |
Тривалість, хвилин |
|
ультразвукова дезінтеграція клітин |
25 |
|
руйнування клітин скляними бусинками |
6 |
|
вплив на клітини мікрохвильового випромінювання |
25 |
|
обробка клітин високою температурою |
20 |
|
фенол-хлороформна екстракція |
150 |
|
|
Скрыпник А. В.
Кандидат ветеринарных наук
Старший научный сотрудник лаборатории молекулярной диагностики
Национальный научный центр «Институт экспериментальной и клинической ветеринарной медицины»
г. Харьков, ул. Пушкинская 83, 61023 Украина |
|



